猴痘病毒CrmB蛋白的结构特征及其抗原表位分析
王 道, 罗洲飞 , 刘 丹
(1.中南大学 湘雅二医院,湖南 长沙 410011;2. 湖南农业大学 生物科学技术学院,湖南 长沙 410128)
猴痘是一种罕见的人畜共患疾病,由猴痘病毒(Monkeypox virus,MPXV)引起。猴痘病毒是一种双链DNA病毒,为非洲西部和中部特有的人畜共患病毒,属于正痘病毒属(Orthopoxvirus,OPXV),该属还包括天花病毒(Variola virus,VARV)和痘苗病毒(Vaccinia virus,VACV)等。1958年,MPXV在丹麦哥本哈根的血清研究所被发现并从灵长类动物分离出来,随后该病毒又在动物园的灵长类动物群落中被实验室人员检测到[1]。1970年首次被确定为人类病原体[2],1980年MPXV成为继VARV后人类致病性最强的OPXV。过去的半个世纪,MPXV在中非和西非的偏远地区以低水平流行,主要由啮齿动物和灵长类动物携带病毒传染给人类[3],现今首次在北美和欧洲广泛传播,引起全球的不安[4]。截止2022年8月9日,全球87个国家和地区猴痘确诊报告病例超过31 800例,美国猴痘确诊人数占全球感染的近30%。2022年7月23日,世界卫生组织(world health organization,WHO)宣布猴痘疫情已构成“国际关注的突发公共卫生事件”。猴痘病毒一般通过接触被病人污染的环境和物品、体液或呼吸道飞沫传播[5],首发临床症状表现为发烧、淋巴结肿、寒战、头痛、肌肉酸痛、背痛和严重疲劳[6],斑丘疹在发烧后1~3 d出现,并逐渐发展成小水泡,然后在大约10 d内结皮。猴痘感染者死亡率为1%~10%,大多数死亡病例为10岁以下的儿童[7]。MPXV的细胞因子应答调节物B(cytokine response modifier B,CrmB)基因存在于病毒基因组的末端反向重复序列中,用来编码一种肿瘤坏死因子(tumor necrosis factor,TNF-α)结合CrmB蛋白。TNF-α是一种由T细胞和巨噬细胞分泌的细胞因子,其保护细胞免受病毒感染的侵袭,杀死已被病毒感染的细胞。CrmB是一种分泌的病毒蛋白,能与TNF-α和TNF-β结合。已有研究表明[8-9],在与宿主长期共同进化的过程中,正痘病毒能够编码由受染细胞分泌的蛋白,结合和阻断宿主免疫系统重要成分的生物活性[10],其中包括肿瘤坏死因子、不同种类的干扰素、白介素-18、趋化因子等。相关文献也报道正痘病毒具有多种免疫逃逸机制:编码E3泛素连接酶和衔接蛋白,直接影响病毒的识别、产生抗病毒信号和炎症[11];生成抑制宿主细胞凋亡的多种Bcl-2模拟物,干扰Bax-Bak途径,使病毒成功感染和复制[12];还包括TNF诱饵受体同系物(viral TNF decoy receptor homologues,vTNFRs)CrmC、CrmD、CrmE[13]、丝氨酸蛋白酶抑制剂CrmA[14]、高尔基抗凋亡蛋白GAAP[15],及双链RNA诱导的抑制凋亡作用的其他蛋白[16]。另外,vTNFRs被发现均具有结合亲和力和特异性的独特特征,以及不同程度的生物活性,由正痘病毒物种差异地表达,表现不同的免疫调节特性[17]。目前,尽管天花疫苗具有一定实质的交叉保护作用[18],人类仍然缺乏针对MPXV特殊治疗的方法或疫苗。此外,MPXV还被部分研究者称有同性传播的可能性[19]。因此,亟需对猴痘病毒CrmB蛋白的结构特性和致病性进行深入了解,才能遏制MPXV流行的蔓延[20]。本文借助多种生物信息学工具探究MPXV CrmB蛋白的理化性质、分子结构和抗原表位等,为我国预防和控制这种新出现的感染猴痘病毒的疫情暴发,加速研发抗MPXV的特异性疫苗和药物提供参考。
1 材料与方法
1.1 材料
通过美国国立生物技术信息中心数据库(national center for biotechnology information,NCBI)搜寻数据并且下载得到MPXV CrmB蛋白的氨基酸序列和核苷酸序列信息,CrmB基因登录号为72551594,编码蛋白登录号为YP_010377181。通过NCBI查询得到该蛋白由349个氨基酸构成,序列如下:MRSVLYSYILFLSCIIINGRDIAPHAPSNG-KCKDNEYRSRNLCCLSCPPGTYASRLCDSKTNTQC-TPCGSDTFTSHNNHLQACLSCNGRCDSNQVETRSC-NTTHNRICECSPGYYCLLKGASGCRTCISKTKCGIG-YGVSGYTSTGDVICSPCGPGTYSHTVSSTDKCEPVV-TSNTFNYIDVEINLYPVNDTSCTRTTTTGLSESISTS-ELTITMNHKDCDPVFRAEYFSVLNNVATSGFFTGE-NRYQNTSKICTLNFEIKCNNKDSSSKQLTKTKNDTI-PMHSETVTLVGDCLSSVDIYILYSNTNTQDYETDT-ISYHMGNVLDVNSHMPASCDIHKLITNSQNPTHL
1.2 方法
采用多种生物信息学工具 (表1),对MPXV CrmB蛋白FASTA格式的氨基酸序列和核苷酸序列进行预测和分析,利用ORF Finder网站预测CrmB基因开放阅读框的数目;运用ProtParam和ProtScale对CrmB蛋白的理化性质与亲/疏水性进行分析;采用SignalP5.0和TMHMM2.0分别分析CrmB蛋白的信号肽和跨膜结构;采用Conserved Domains和Cell-Ploc2.0预测CrmB蛋白的结构域以及其在宿主细胞的亚细胞定位;运用SOPMA预测蛋白的二级结构,SWISS-MODEL预测蛋白的三级结构;运用NetPhos3.1和NetNGlyc1.0预测磷酸化位点和糖基化修饰位点;通过IEDB筛选CrmB蛋白的线性优势B细胞表位;通过SYFPEITHI对CrmB蛋白的优势T细胞表位进行分析;运用Clustalx软件比对MPXV CrmB蛋白与其他物种的相似性;利用本地MEGA 11.0软件构建进化树。

表1 重要生物信息数据库和网站Table 1 Important bioinformatic databases and websites
2 结果与分析
2.1 CrmB基因的开放阅读框
ORF Finder 在线网站输入 MPXVCrmB基因的核苷酸序列进行预测,结果表明,CrmB基因位于全基因组(NC_063383.1)的194 459~195 508位,总长度为1 050 bp,CrmB基因总共包含10个开放阅读框,ORF1是最长的开放阅读框,起始密码子为“ATG”,终止密码子为“TAA”,编码349个氨基酸,与目的蛋白全长一致,证明CrmB蛋白全长可以完全表达(图1)。

图1 CrmB 基因的开放阅读框分析Fig.1 Open reading frame analysis of CrmB gene
2.2 CrmB蛋白的理化性质和亲/疏水性
进入ExPasy网站,利用ProtParam在线对MPXV CrmB蛋白的理化性质分析,结果表明,该蛋白的相对分子量为38 308.75,理论等电点为6.24,分子式为C1621H2550N460O550S32,分别含有27个带正电荷的氨基酸残基 (Arg+Lys) 和31个带负电荷的氨基酸残基 (Asp+Glu),总原子数5 213,消光系数26 955,半衰期30 h,不稳定系数42.90,属于不稳定蛋白,脂肪系数为64.47,亲水性平均值为-0.403。组成TNF受体蛋白的氨基酸共有19种,其中含量最高的为丝氨酸,占12.0%;含量最低的为甲硫氨酸,占1.4%,不含色氨酸、吡咯赖氨酸和硒代半胱氨酸 (表2)。
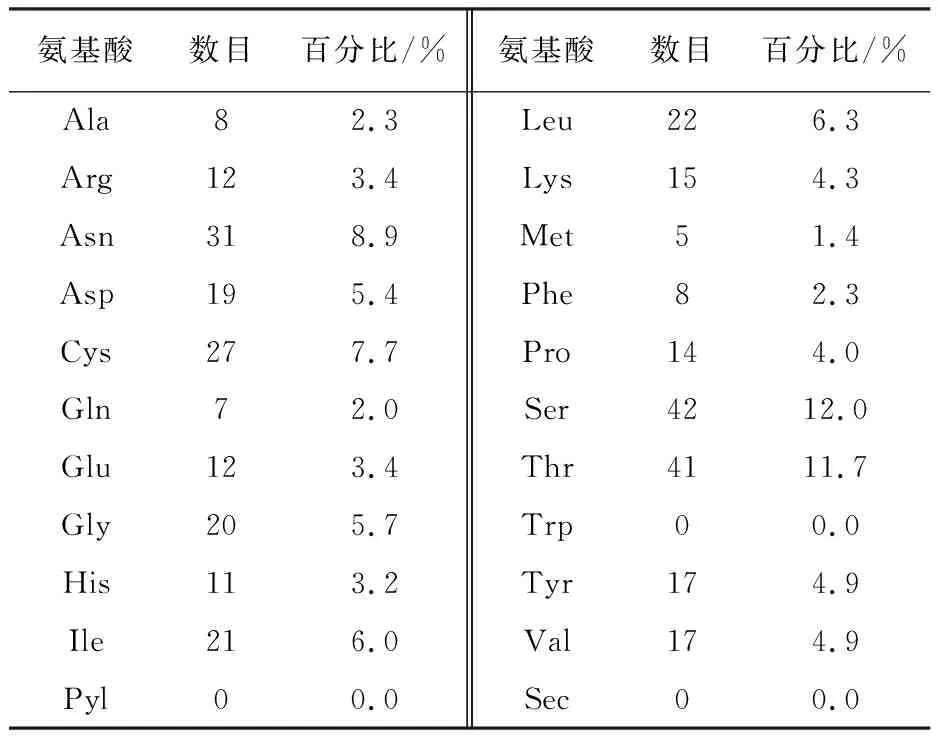
表2 CrmB蛋白的氨基酸组成Table 2 Amino acid composition of CrmB
利用ProtScale在线分析CrmB蛋白的亲疏水性,结果表明,最强的亲水位点在第37位的酪氨酸,亲水值为-3.222;最强的疏水氨基酸是13位的丝氨酸,亲水值为3.344。亲水性氨基酸残基数目比疏水性氨基酸残基多,推测该蛋白属于亲水性蛋白 (图2)。
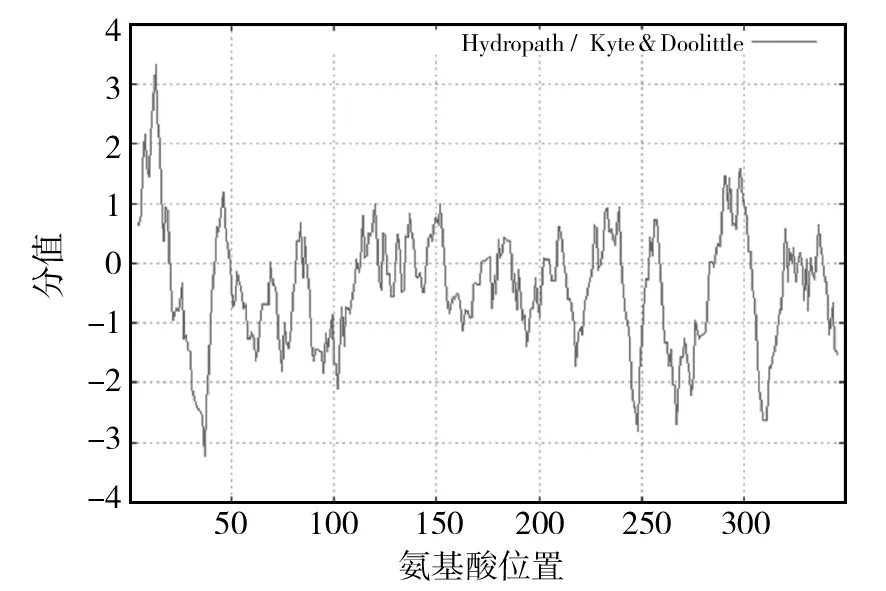
图2 CrmB蛋白的亲/疏水性分析Fig.2 Hydropathicity/Hydrophobicity analysis of CrmB
2.3 CrmB蛋白的信号肽结构、跨膜结构及结构域
登录SignalP 5.0 Server网站,分析CrmB蛋白信号肽序列,结果显示,存在信号肽的概率为0.997 67,大于阈值0.5,表明该蛋白是分泌型蛋白,信号肽切割位点位于G19~R20之间,概率为0.836 9,信号肽序列可能为“MRSVLYSYILFLSCIIING”(图3)。TMHMM 2.0网站预测CrmB蛋白不具有跨膜结构 (图4)。Conserved Domains 预测结果表明,CrmB蛋白在32~154位氨基酸序列存在TNFRSF超家族结构域,该家族包含病毒TNFR同源物,有助于TNF介导的抗病毒过程的修饰;在174~334位氨基酸序列存在DUF1406超家族结构域,该家族由几种长度约为185的正痘病毒蛋白组成,一些家族成员被注释为C8蛋白,但功能未知(图5)。
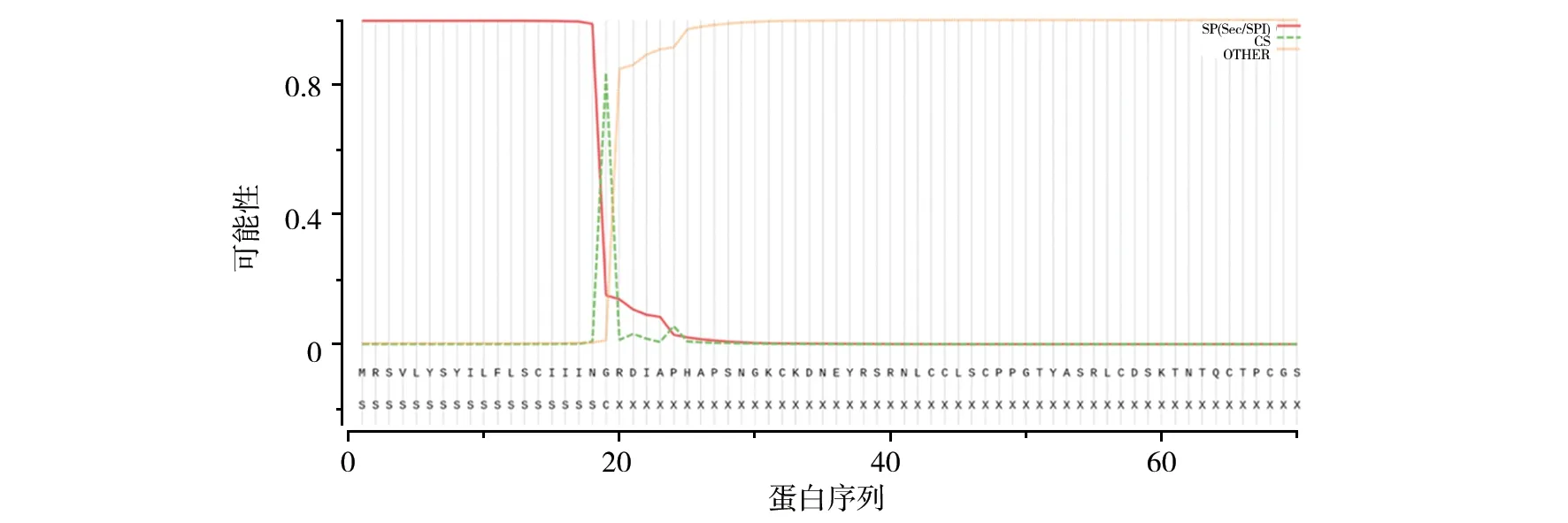
图3 CrmB蛋白的信号肽预测Fig.3 Signal peptide prediction of CrmB
图4 CrmB蛋白的跨膜结构预测Fig.4 Transmembrane domain prediction of CrmB
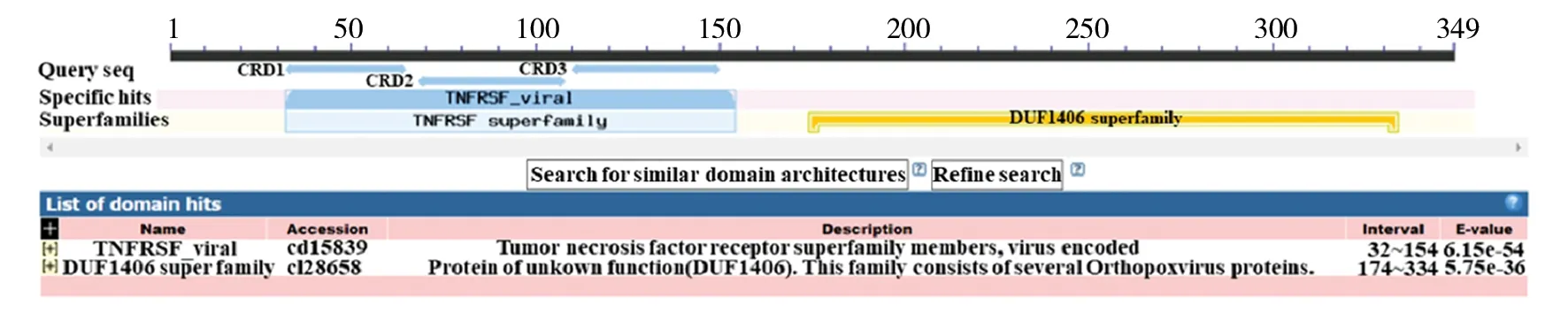
图5 CrmB蛋白的结构域预测Fig.5 Conserved domain prediction of CrmB
2.4 CrmB蛋白的二级结构和三级结构
SOPMA在线网站预测CrmB蛋白质的二级结构。结果显示,多肽链中α-螺旋占4.87%,延伸片段占30.66%,无规则卷曲则占57.59%,β-转角占6.88%,无规则卷曲所占比例最高(图6)。运用SWISS-MODEL构建出4个同源模型,然后根据GMQE和QMEAN数值的大小选取最为可靠的CrmB蛋白的三级结构模型A,其GMQE分值为0.36,QMEAN分值为0.76。利用拉氏图验证蛋白三级结构模型的可靠性,模型A拉氏图表明,90.1%的氨基酸残基位于最合理区,9.3%的氨基酸残基位于其他合理区,说明形成的二面角合理,推测模型A具有较好的可信度(图7)。
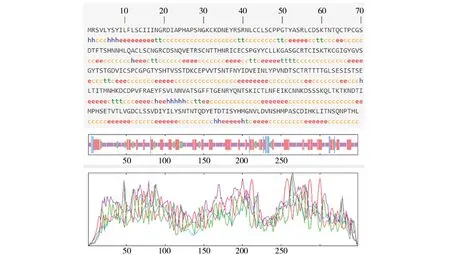
图6 CrmB蛋白的二级结构Fig.6 Secondary structure protein of CrmB
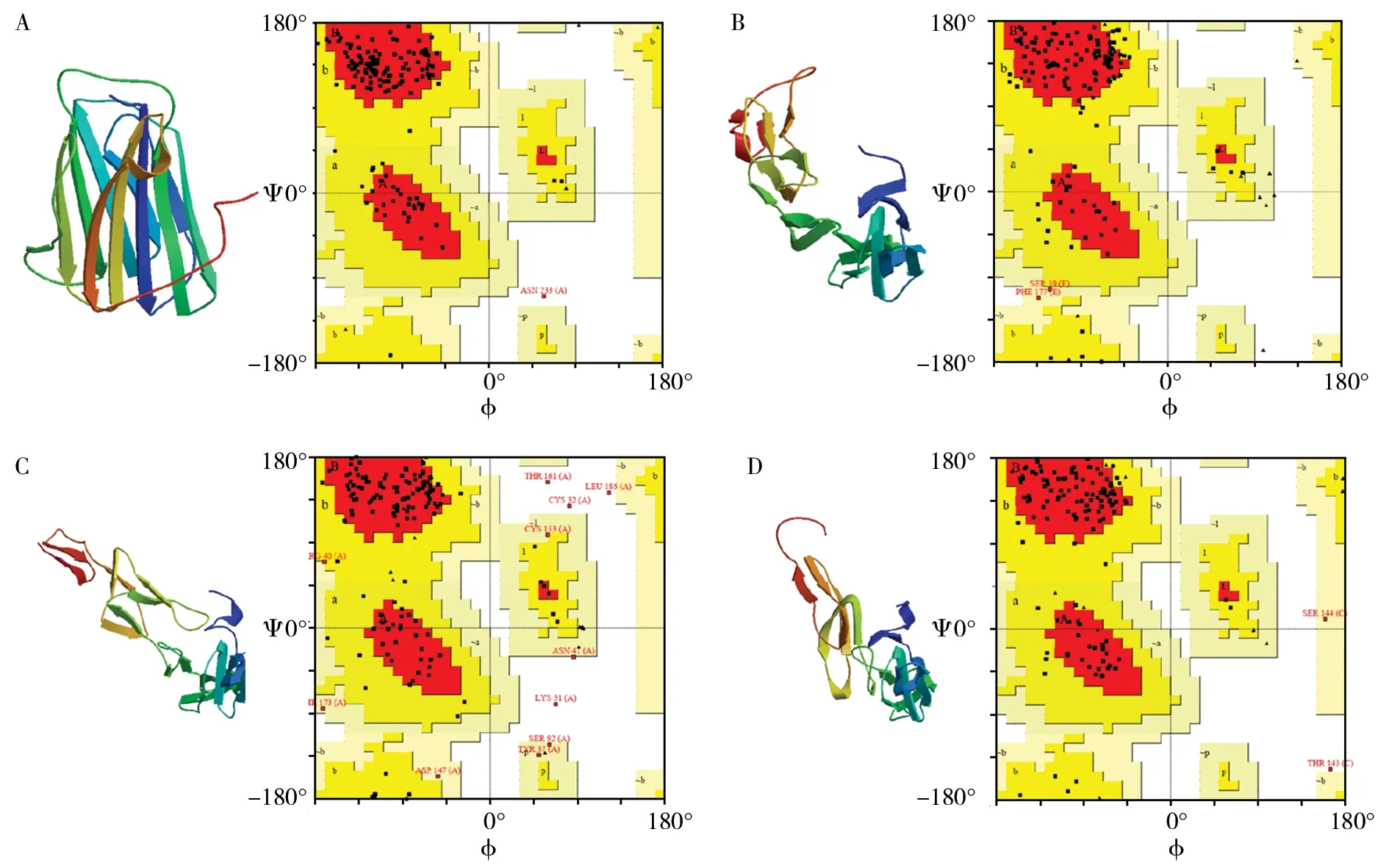
图7 CrmB蛋白的三级结构Fig.7 Tertiary structure protein of CrmB proteinA:模型A;B:模型B;C:模型C;D:模型DA:Model A;B:Model B;C:Model C;D:Model D
2.5 CrmB蛋白磷酸化和糖基化位点及亚细胞定位
进入ExPasy网站,利用NetNGlyc1.0、NetPhos3.1在线分别对CrmB蛋白N-糖基化和磷酸化位点进行分析。结果表明,CrmB蛋白存在6个潜在的N-糖基化位点,分别位于第101、190、249、277、313和345位的氨基酸,其中101(63.64%)、190(54.41%)、249(66.51%)和277(56.31%)位点的糖基化概率较高 (图8);设定阈值为0.5时,CrmB蛋白具有 54个潜在的磷酸化位点,其中丝氨酸磷酸化位点27个,苏氨酸磷酸化位点20个,酪氨酸磷酸化位点7个 (图9),全部磷酸化位点和对应激酶见表3。利用Cell-Ploc2.0 预测CrmB蛋白的亚细胞定位,结果显示主要在宿主细胞的细胞质膜上发挥作用(图10)。
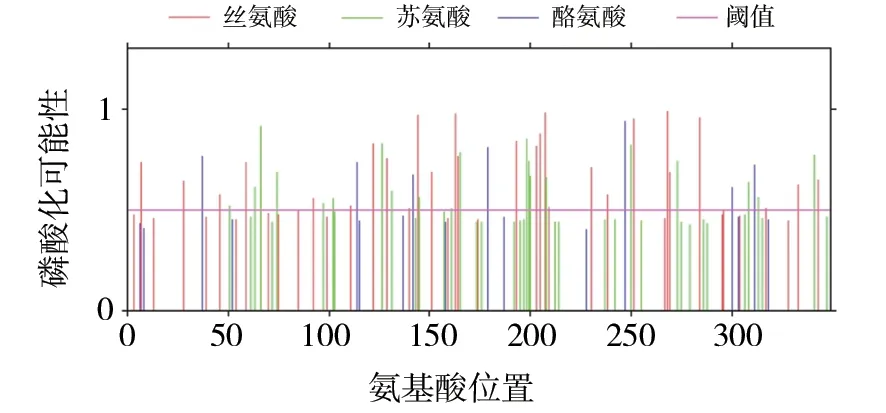
图9 CrmB蛋白磷酸化位点预测Fig.9 Phosphorylation sites prediction of CrmB
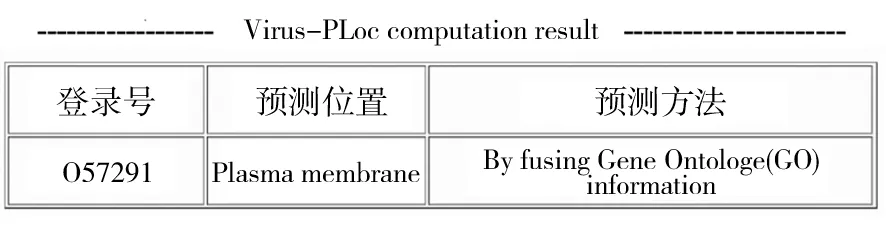
图10 CrmB蛋白亚细胞定位预测Fig.10 Subcellular localization prediction of CrmB
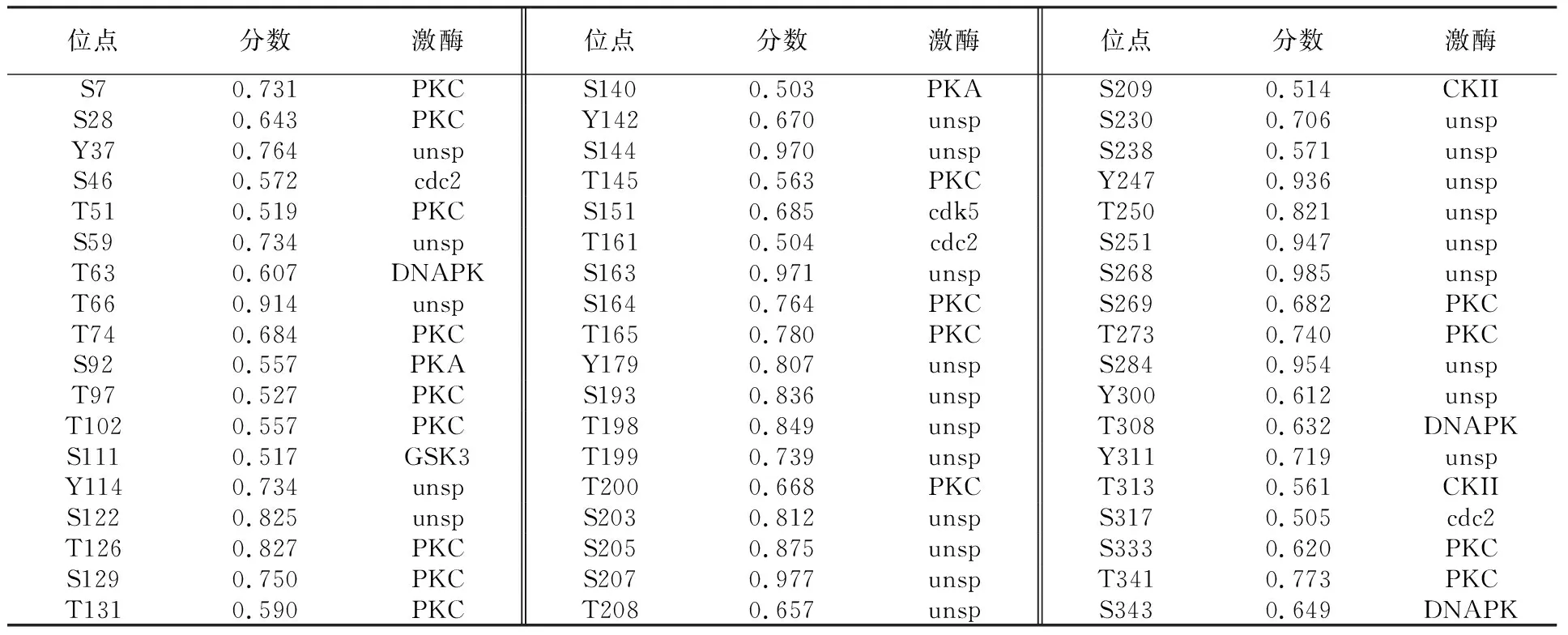
表3 CrmB蛋白的磷酸化位点和对应激酶Table 3 Phosphorylation sites and corresponding kinases of CrmB
2.6 CrmB蛋白的B细胞表位分析
进入IEDB在线网站,预测CrmB蛋白的优势B细胞表位,阈值为0.5。CrmB蛋白共有8个线性B细胞抗原表位,分别为R19~C43、G49~T62、S69~I106、Y114~I148、G155~T287、T305~T314、L323~C333、H336~T346 (图11)。
2.7 CrmB蛋白的T细胞表位分析
运用SYFPEITHI网站,对CrmB蛋白的T细胞表位进行预测分析,其中MHC I类分子亚型 HLA-A*02∶01限制性CTL表位,分值在20以上,检测出8个CTL细胞优势表位(表4);MHC II类分子亚型HLA-DRB1*04∶01辅助性Th细胞表位,分值在22以上,检测出13个Th细胞表位(表5)。
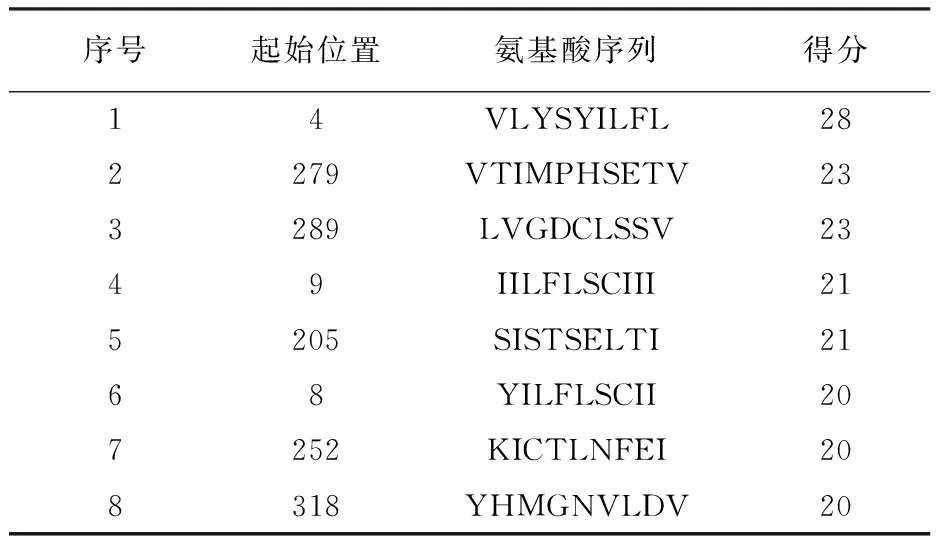
表4 CrmB蛋白CTL细胞优势表位分析Table 4 Analysis of dominant epitopes of CrmB in CTL cells
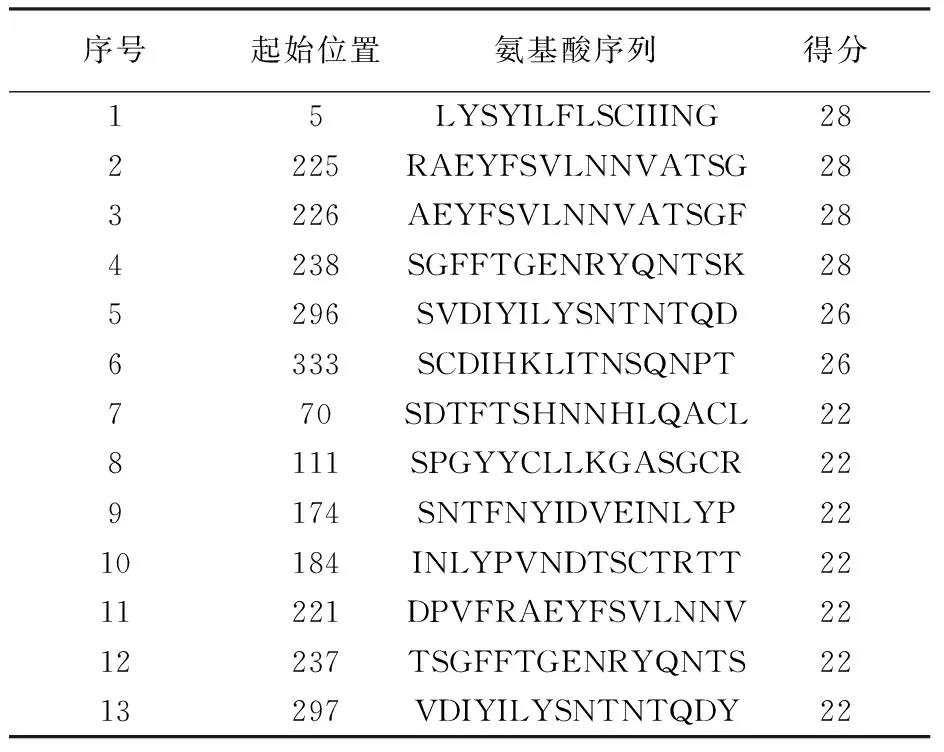
表5 CrmB蛋白Th细胞优势表位分析Table 5 Analysis of dominant epitopes of CrmB in Th cells
2.8 CrmB蛋白的抗原决定簇分析
采用Immunomedicine Group在线网站对CrmB蛋白的抗原决定簇预测分析,平均抗原倾向性为1.034 4,总共有15个抗原决定簇表位,主要位于氨基酸序列的4~16、39~50、52~58、64~70、77~88、104~141、144~155、157~164、166~173、178~191、218~234、250~256、283~303、319~328、330~340位点(图12),表明该蛋白抗原倾向性高,容易形成抗原决定簇。
2.9 CrmB蛋白的配体结合位点和小分子药物分析
基于随机森林算法,利用Prankweb平台预测CrmB蛋白表面上的配体结合位点。图13A的序列可视化中,5种颜色的矩形分别代表预期蛋白质口袋结构和实际配体相结合的可能区域。图13B的结构可视化中,蛋白质表面用不同颜色表示各个口袋区域,灰色代表保守性区域,灰色越深表示保守性越高。表6的数据显示,蓝色区域 (Pocket 1) 总分值是5.76,排名为1,大于其他颜色区域的总分值,说明蓝色区域是最大可能的配体结合区域,其平均可能性得分为0.297,表面氨基酸残基数目为12,平均保守性得分2.702。同时,基于药物-基因组学,利用DrugBank数据库预测小分子药物。结果显示,存在两种小分子药物可能能够抑制CrmB蛋白(数据库登录号分别为DB05959和DB03507),其化学式如图14所示。
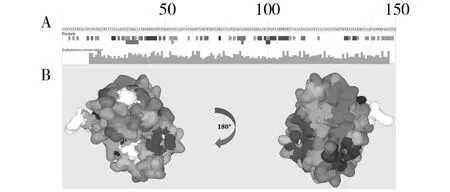
图13 CrmB蛋白的配体结合区域分析Fig.13 Prediction of ligand binding sites of CrmBA:进化同源性;B:蛋白-配体结合位点A:Protein evolutionary;B:Protein-ligand binding site
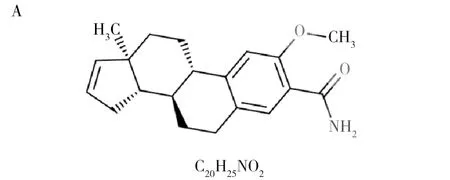
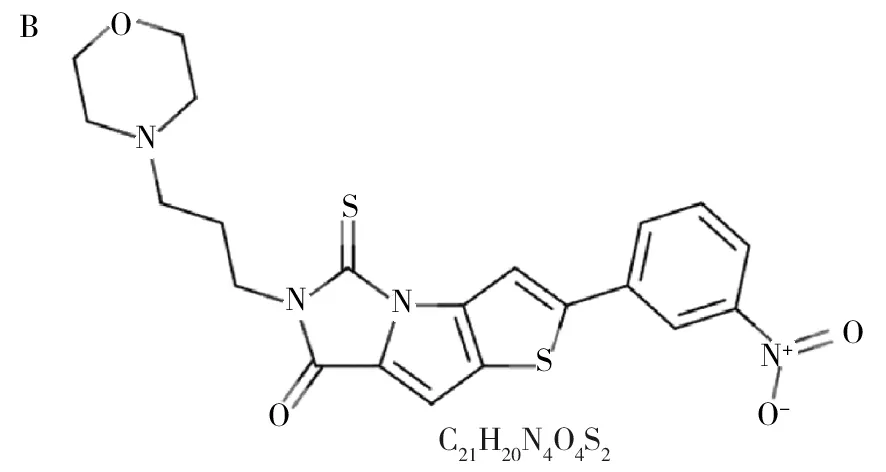
图14 CrmB蛋白的小分子药物预测Fig.14 Prediction of small molecular drugs of CrmBA:2-甲氧基雌激素-1,3,5(10)1,6-四烯-3-甲酰胺;B:2-(3-硝基苯基)-4-硫氧基-5-(3-吗啉丙基)-5,6-二氢-1-硫-3b,5-二唑-4H-环戊二烯-6-酮A:2-methoxyoestra-1,3,5(10)1,6-tetraene-3-carboxamide;B:2-(3-Nitrophenyl)-4-thioxo-5-(3-morpholinopropyl)-5,6-dihydro-1-thia-3b,5-diaza-4H-cyclopenta[a]pentalen e-6-one

表6 CrmB蛋白的配体结合区域数据Table 6 Datas of ligand binding sites of CrmB
2.10 CrmB蛋白在不同物种中的序列比较和同源性分析
运用Uniprot数据库获取不同物种CrmB蛋白的氨基酸序列,Clustalx对其进行同源性比对分析 (图15)。从CrmB蛋白同源性矩阵可以得出,猴痘病毒 (Monkeypox virus,MPXV)与猫痘病毒 (Feline poxvirus,FPXV)、天花病毒 (Variola virus,VARV)、骆驼痘病毒 (Camelpox virus,CMLV) 以及牛痘病毒 (Cowpox virus,CPXV) 蛋白序列的同源性分别为87.86%、85.30%、89.91%、91.35% (图16)。进化树结果显示,猴痘病毒与猫痘病毒亲缘关系最近,其次是天花病毒,牛痘病毒和骆驼痘病毒构成一小分支与猴痘病毒距离较远 (图17)。

图16 不同物种CrmB蛋白的百分比同源性矩阵Fig.16 Percent identity matrix of CrmB in different species
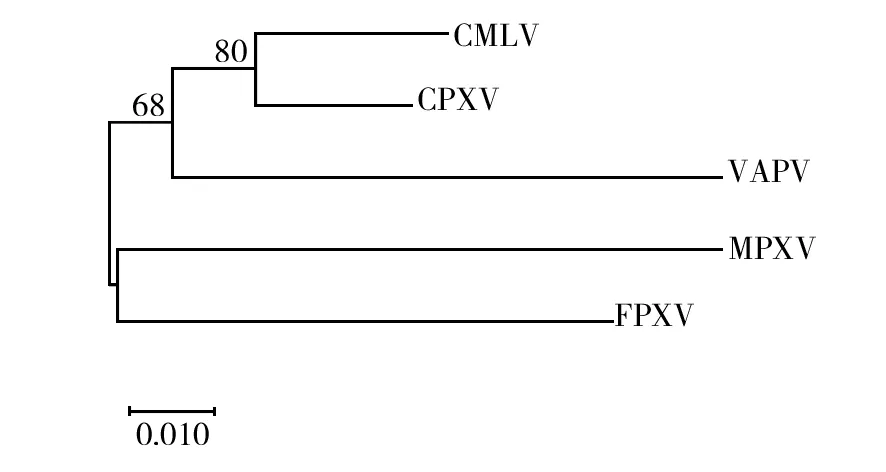
图17 不同物种间CrmB蛋白的进化树Fig.17 Phylogenetic tree of CrmB in different species
3 讨 论
现今,国内继香港发现首起猴痘病例以来,重庆又出现了一起境外输入猴痘病例。但由于这次MPXV的特点和未知的风险,特别是中国人口众多,大规模传播的风险并不能完全排除[21]。我们应加强对MPXV的基础研究,利用生物信息学工具快速追踪病毒源头,识别病毒和宿主之间的相互作用,分析包括开放阅读框、同源性、序列比对、糖化位点、蛋白二级和三级结构、抗原表位、配体结合区域等,这是短时间内找到预防和治疗MPXV最有效的方法。
本研究比对结果显示MPXV和VARV、CPXV CrmB氨基酸序列同源性分别为85.30%、91.35%,说明三者CrmB蛋白相似程度较高,并且进化树也表明三者亲缘关系较近。所以,MPXV引起人类疾病的症状类似VARV,但没有VARV那么严重。相关文献报道CPXV是VARV、MPXV、VACV、CMLV祖先最近的亲属[22]。虽然它们有一个共同的祖先,但MPXV不是VARV的直接祖先或者后代。以往研究表明,虽然VARV和MPXV在遗传上和抗原上是相关的,但是两者基因组的毒力因子和宿主范围编码区域内的序列是不同的[23]。所以,尝试VARV作为MPXV替代模型,特别是在疫苗检测和治疗对策方面,基因组的差异可能导致不同宿主的发病机制明显不一样。而且,2022 MPXV基因组与2017/2019 MPXV相比较,鉴定存在47个单核苷酸变异,对于MPXV在各国传播也还没有明确的预防方法,仅天花疫苗被证明对预防MPXV有85%的有效率[24]。
本研究发现MPXV CrmB蛋白含有一段约为20个氨基酸的信号肽序列,但不存在跨膜结构区域,提示该蛋白在宿主细胞合成转运时信号肽并没有被清除。信号肽是细胞膜靶标和膜插入的关键因素,在其他种类病毒如乙型脑炎病毒[25]、HIV-1[26]、丙型肝炎病毒[27]都有报道,推测此信号肽可能具有定位作用,有利于CrmB蛋白在宿主体内稳定合成且促使子代病毒复制。若将信号肽进行阻断干扰,影响蛋白的正常功能,进而使子代病毒不能合成复制,不失为一种遏制病毒大量传播的新途径。
从MPXV CrmB蛋白的结构上分析,该蛋白的二级结构以无规则卷曲为主,结构域分析表明属于TNFRSF 超家族。因为无规则卷曲结构的存在,使蛋白空间结构容易发生变化,有利于与其他物质结合。三级结构模型A具有较好的可靠性,因为拉氏图显示90.1%氨基酸残基点在深红色区域和9.3%氨基酸残基点在深黄色区域。同时,模型A三级结构的β-折叠 I由5条大的反平行链组成,而β-折叠II也由5条链组成。这些结果都与Nelson等[28]报道正痘病毒属编码的蛋白质采用特征性的β-夹心折叠相一致。理化性质还表明,MPXV CrmB蛋白在哺乳动物中的半衰期为30 h,不稳定系数为42.90,脂肪系数为64.47,属于不稳定蛋白,说明该蛋白更容易折叠,形成一定的空间结构,这可能与猴痘病毒入侵宿主的方式相关。另外,CrmB蛋白还是一种不稳定的亲水性蛋白,相关文献证实该蛋白是一种可溶性蛋白质,拥有TNFRSF胞外结构域,能与溶液中的肿瘤坏死因子配体超家族 (TNF ligand superfamily,TNFLSF) 成员结合,阻碍其与细胞表面中的同源受体相互作用,从而反向信号转导抑制炎症[29]。
蛋白糖基化具有广泛的作用,包括介导蛋白质折叠和稳定性、免疫逃逸、宿主受体附着和细胞进入[30-31]。同样,蛋白磷酸化也是调节蛋白功能最普遍机制[32-33]。本研究发现CrmB蛋白可能存在6个糖基化位点、54个磷酸化位点、5个配体结合区域以及2个小分子抑制药物,这些结果都将为药物设计提供参考。美国药监局已经批准抗病毒口服药tecovirimat作为首选治疗人类猴痘,但其安全性还有待大规模数据观察[34]。对比其他两种药物brincidofovir和cidofovir,前者安全性可能较好,但在动物模型中的研究较少[35]。
虽然大多数猴痘患者症状轻微或具有自限性,但先前文献报道注射免疫人痘疫苗可能具有一定的保护效应和改善临床感染症状[36]。本研究通过IEDB网站分别对CrmB蛋白的B细胞线性表位进行了分析,筛选出8个最可能的优势抗原表位;通过 SYFPEITHI 网站依据分值筛选出CTL细胞最可能的优势表位有8个,Th细胞优势表位有13个;Immunomedicine Group网站也证实CrmB蛋白容易形成抗原决定簇,这些结果为研发特异性强、副反应小、安全性高的疫苗提供了可能性,以替代现今美国和欧洲批准的人用天花疫苗JYNNEOS[37]和ACAM2000[38]。
MPXV与仍在流行的SARS-CoV-2有着很大的不同,MPXV基因组非常庞大,比SARS-CoV-2大6倍以上[39]。现今,MPXV在自然界中的死灰复燃是否会替代新冠病毒的流行病学地位仍然不确定。本研究借助生物信息学手段迅速分析了MPXV CrmB蛋白结构,预测了配体结合位点和抗原表位,为加快疫苗和药物研发提供参考。

