放线菌来源活性天然产物发现研究进展
尚 坤, 阎瑾逸, 高君梅, 王 瑾, 李思颖, 闫祥祥, 黄小玉, 刘 晨, 岳昌武
(延安大学基础医学院 延安市微生物药物创新及转化重点实验室,陕西 延安 716000)
微生物来源的抗生素类药物在临床治疗病原菌感染和肿瘤等重大疾病方面发挥着重要作用,是抗生素的主要来源,具有非常好的临床应用前景(表1)[1]。由于近年来抗生素的滥用等原因导致临床上出现的多药耐药甚至全耐药菌株的数量和种类急剧增加,从2010年“超级菌NDM-1”全球火速扩散到2015年2月美国UCLA医疗中心爆发“超级菌”肆虐,这些耐药超级菌严重威胁着人类的生命健康,已成为全球医疗卫生领域面临的一道亟待攻克的难题。抗生素权威Walsh曾指出,超级菌可让致病菌变得无比强大,抵御几乎所有抗生素,而且由于新抗生素的研发、上市速度严重滞后,可能会导致“超级菌”感染,10年内将无药可用[2]。由于细菌耐药给全球临床抗生素用药带来了巨大挑战,近年来,新抗生素的研发重新受到医药行业和各国政府高度重视。自21世纪以来,基因组学、代谢组学以及基因组编辑等技术的巨大进步,促使科学家们开始反思和改进抗生素研发策略,提出了许多诸如基于分子系统学、代谢组学以及基因组挖掘技术的新抗生素发现新策略,并取得了诸多进展。本文总结了20世纪起从放线菌中发现的重要活性天然产物及其策略和方法,对新时期放线菌来源抗生素的开发现状、放线菌新菌株的筛选、放线菌来源天然产物快速鉴定、沉默基因簇等基因组挖掘的技术方法等进行综述,以期为放线菌来源的活性天然产物的发现提供借鉴。

表1 已知天然产物及其合成类似物数量及应用前景Table 1 Quantity and application prospect of natural products and their synthetic analogues
1 放线菌来源活性天然产物的开发现状
放线菌来源的活性天然产物是抗生素的重要来源,目前发现放线菌来源的微生物生物活性物质超过20 000个,其中抗生素活性天然产物超过13 800个,除了目前还在临床上广泛应用的链霉素、卡那霉素、氯霉素等抗生素外,还包括免疫调节剂、受体拮抗剂以及酶、维生素、有机酸等生物活性物质[1]。按照微生物天然产物开发历程,可将放线菌天然产物的发现分为三个阶段[3]:前30年(1940年代至1970年代)、第二个 30 年(1970 年代至 2000 年代)和2000 年代及以后。从表2可以看出,尽管进入新时期从普通生境中分离出新的放线菌种以及挖掘新的活性天然产物已变得越来越困难,放线菌来源的活性产物在微生物来源活性产物中的比重也在不断降低(从早期的62%降到了目前的28.5%)。科学家们基于“新环境,新菌株,新产物”策略,从特殊生境或稀有放线菌中通过各种基因组挖掘等方法开发新型抗生素,并取得了积极成效。据Kemung等[4]统计,从1994年到2017年,通过链霉菌抗生素发现新策略,已从各种生境链霉菌中发现至少150种以上具有抗耐甲氧西林金黄色葡萄球菌(methicillin-resistantStaphylococcusaureus, MRSA)活性的药物前导化合物,提示这不失为开发放线菌来源抗生素的重要途径。
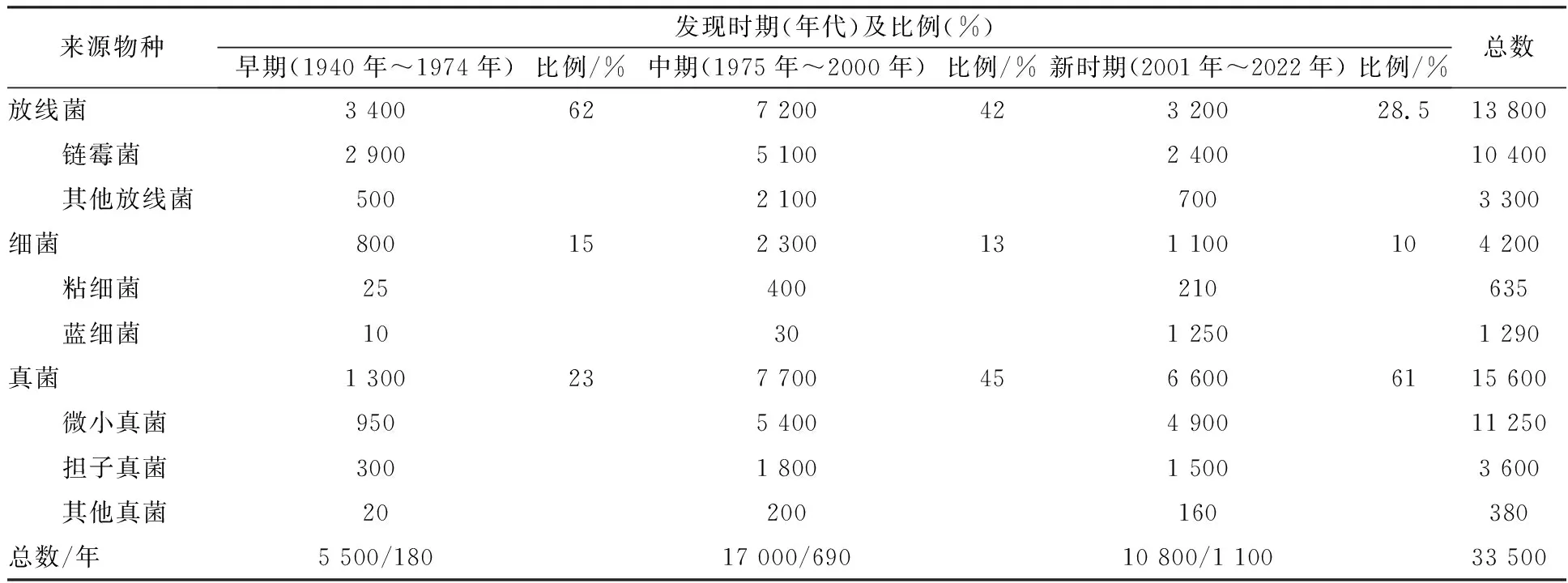
表2 1940年至2022年间报道的微生物活性产物的大致数量及比例[1]Table 2 Quantity and proportion of microbial active products reported from 1940 to 2010[1]
2 新时期典型生境放线菌来源活性天然产物的发现
进入21世纪以来,以高分辨质谱等为代表的现代质谱技术、代谢组学为代表的现代组学技术的巨大进步以及上述天然产物开发策略的调整,发现了一大批新型放线菌来源的抗生素。
2.1 大环内酯类化合物
周啟辉[5]从高纬度北海海洋中分离出链霉菌(Streptomycessp.) R-527,并从中分离出9个(P1~P9)活性化合物,其中化合物P1~P4是大环内酯类化合物,P5、P7、P8为开环类化合物。Li等[6]从东南极洲潮间带沉积物中分离的稀有放线菌海沉淀菌(Marisediminicola) ZS314T中分离出具有很高抗菌活性的萜类、烷基间苯二酚、低聚糖或氨基酸衍生的大环内酯类系列化合物[7]。
2.2 新型大环多烯内酯类化合物
刘洋等[8]从威海近海海域分离出链霉菌(Streptomycessp.) B59菌株,运用OSMAC(one strain-many compounds,一株菌株,多个产物)方法挖掘出1种具有独特环戊烯酮结构存在的新型大环多烯内酯类化合物B59-1,经初步测试,化合物B59-1在50 μg/mL时抑制稻瘟病菌孢子萌发,抑制率可达到100%。
2.3 生物碱类化合物
矫文策[9]从大连周边海域分离的星海链霉菌(Streptomycesxinghaiensissp. nov.)中挖掘出具有良好抑制肿瘤细胞活性的含亚砜官能团的生物碱类次级代谢产物Xinghaiamine A。 周彪[10]分别从链霉菌(Streptomycessp.) DT-A56和链霉菌(Streptomycessp.)NB-A13的大米固体培养基和高氏液体培养基的培养物中分离出具有双吲哚化学结构的生物碱星孢菌素类化合物,其中从菌株DT-A56培养物中分离出15个化合物(化合物1~15),其中3个为新化合物(化合物1~3);从菌株NB-A13培养物中分离出19个化合物(化合物16~34),其中6个为新化合物(化合物16~21)。这些新化合物对前列腺癌和乳腺癌的治疗具有潜在的应用价值。
2.4 酰胺类化合物
孙金柱[11]从链霉菌(Streptomycessp.)strain nenu_DS_21分离出10个化合物(Ⅰ~Ⅹ),其中Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅶ、Ⅸ化合物为苯甲酰胺类化合物,Ⅴ、Ⅵ、Ⅶ为首次从自然界中分离出的酰胺类天然产物,Ⅹ号化合物为新的链型酰胺类化合物。该放线菌主要的次级代谢产物是化合物Ⅷ。抗菌实验结果显示,化合物Ⅷ对番茄灰霉病菌(Botrvtiscinerea)、水稻纹枯病菌(Rhizoconiasolani)、苹果斑点落叶病菌(Pseudomonassp.)有一定的抑菌活性。
2.5 聚酮类化合物
Lü等[12-13]从梵净山原始森林土样中分离的链霉菌(Streptomycessp.) FJS31-2中得到发酵天然产物zunyimycins化合物(图6),该系列抗生素对MRSA和肠球菌有较强的杀菌作用,还可抑制肝母细胞瘤、肺癌以及人胃癌细胞、黑色素瘤等多种癌细胞株的增殖,对抗肿瘤新药的开发具有重要的意义[14]。Wyche等[15]从来自海洋的马杜拉放线菌(Actinomadurasp.)WMMB-499中分离出了新型低毒抗真菌聚酮Forazoline A。
2.6 噻嗪类化合物
Fu等[16]从海洋来源放线菌绿藻(Actinomycetosporachlora) SNC-032中分离出了一种新的噻嗪和两种新的噻唑衍生物,并将其命名为Thiasporines A~C。其中Thiasporines A具有独特的5-羟基-2-苯基-4h-1,3-噻嗪-4-1核。Thiasporines A对非小细胞肺癌细胞H2122表现出中度的细胞毒作用。
2.7 环肽类化合物
郭俊夫[17]从海南岛沿海浅海区生长的菌株绿色链霉菌(Streptomycesviridobrunneus) P10-16中纯化出系列具有抗细菌、抗真菌、抗病毒、抗肿瘤等生物活性的环脂肽类的伊枯草菌素(Iturin A)类化合物GJF6-2、GJF6-3q、GJF6-3h、GJF6-4q以及GJF6-4h,这类化合物的疏水部分是由14~17个碳原子组成的β-氨基脂肪酸,亲水部分是肽链,是由7个α-氨基酸通过由脂肪酸的氨基与C-末端氨基酸的羧基形成的酰胺键成环典型结构[18]。
2.8 放线菌素类化合物
天然的放线菌素类化合物的发色团母核均为吩噁嗪酮,即2-氨基-4,6-二甲基吩噁嗪-3-酮-1,9-二羧酸。2011年,王强[19]从链霉菌(Streptomycessp.)IMB094和糖单孢菌(Saccharomonosporasp.) 7-98-1菌株的活性次级代谢产物中分离出4个放线菌素类化合物(化合物1~4),其中化合物1和2的生色团母核为罕见的噁唑并[4,5-b]吩噁嗪四环稠合结构,这种母核结构在天然放线菌素中首次被发现。化合物1为新结构化合物,命名为新放线菌素A。化合物2为首次发现天然放线菌素衍生物,命名为新放线菌素B。通过其抗菌活性测试结果显示,新放线菌素A可以对耐甲氧西林金黄色葡萄球菌、甲氧西林敏感金黄色葡萄球菌(methicillin-susceptibleStaphylococcusaureus)、甲氧西林耐药的表皮葡萄球菌(methicillin-resistantStaphylococcusepidermidis)等多种耐药菌株具有中等的抗菌活性。并且对肺腺癌、结肠癌、肝癌等癌细胞具有较强的抗肿瘤细胞活性。
2.9 糖苷类化合物
保罗霉素(Paulomycin) 是对金黄色葡萄球菌(Staphylococcusaureus)和蜡样芽胞杆菌(Bacilluscereus)等革兰阳性细菌具有很强活性的抗生素,用于治疗淋球菌和衣原体感染[20]。Sarmiento-Vizcaino等[21]在海底Avilés峡谷进行海洋考察时,从2 000 m的深海沉积物中分离出来的松木小单孢菌M-412中,分离出了一种没有报道过的新的 Paulomycin G,该抗生素是一种新型的天然产物,结构独特,它是Paulomycin家族中第1个缺乏Paulomycase部分的抗生素,是报道的最小的生物活性的Paulomycin。该天然产物是第一个对不同的人类肿瘤细胞系,如对胰腺癌、乳腺腺癌和肝细胞癌的家族成员表现出强烈的细胞毒活性,Paulonycin G值得考虑作为进一步研究其抗癌潜力的候选药物。
3 激活次级代谢基因表达促进抗生素合成
放线菌是抗生素的重要来源,其次级代谢途径编码基因表达与否直接决定了该次级代谢产物能否合成。Borodina等[22]分析了天蓝色链霉菌(Streptomyxescoelicolor)A3(2)的近700个代谢相关基因在不同生理状态下的表达状态,发现约有58%的代谢相关基因只有在特定环境状态下才被诱导表达。如何有效激活“沉默”的次级代谢基因,最大程度发掘菌株的次级代谢潜力已成为当前新抗生素开发研究人员面临的重要课题。
目前针对放线菌的沉默基因激活主要有两种策略,一种是在生理层面改变放线菌的生活环境,主要有混合培养、OSMAC、化学表观遗传调控、核糖体工程等策略。另一种是在基因层面对基因的直接操作,主要包括调控基因的遗传操作、基因簇倍增以及异源表达等策略。
3.1 基于生理层面改变放线菌活性天然产物开发
混合培养是基于自然生态环境中的微生物往往处于多菌种共存的状态,模拟微生物野生生境,将分离菌株和MRSA共培养,可能有利于增加次生代谢产物的多样性及新生物活性物质的发现机会。OSMAC策略是通过改变培养基营养成分、pH值、含氧量、温度、培养方式等随机地激活体内相关基因表达,继而改变菌株的代谢途径来产生新的次级代谢产物或提高其产量,该策略简单有效,是目前微生物次级代谢产物开发的重要手段。化学表观遗传学(Chemical epigenetics)是向培养基中添加外源性可影响菌株次级代谢途径调控的胞外小分子化合物信号分子或DNA甲基化酶、去甲基化酶抑制剂、拓扑异构酶抑制剂等,以改变染色质结构或调控其次级代谢基因簇转录活性的化学物质[23],启动或抑制菌体内的相关代谢通路,从而实现与体内基因表达水平调控相同或相似的生物学效果。核糖体工程(Ribosome engineering)选用作用于核糖体的抗生素,通过菌株对相应抗生素亚致死量的抗性等来筛选得到代谢途径或代谢产物合成效率得到改造的突变株。核糖体工程可用于定向筛选获得耐药突变株,进而检测这些耐药株次级代谢能力的改变。以链霉素为例,链霉素作用靶点为细菌的核糖体组分,当受到外界高浓度链霉素的选择,部分菌株核糖体蛋白发生突变,成为耐药菌株,而这些核糖体蛋白组分的突变往往能刺激或提高菌株内部抗生素的生物合成[24]。基因的遗传操作步骤较繁琐,需要“因菌而异”地优化遗传操作条件,以适合于对遗传背景较清楚的潜力菌株进行代谢潜力挖掘。而混合培养、OSMAC、表观遗传调控等天然产物挖掘策略,由于其操作便捷,则适合于对遗传背景并不太清楚的菌株资源库进行大规模、高通量挖掘。因此,如果能够通过提供一个较为理想的发酵条件或通过在培养基中添加相应的成分等手段,激活放线菌基因组中常规条件下处于沉默状态的抗生素合成隐性基因,就可能有效地启动其编码的次级代谢产物合成,这将是获得新型放线菌来源抗生素的重要途径,这一观点已得到大量研究结果的证实[25],延安市微生物药物创新及转化重点实验室曾利用基因组挖掘技术结合OSMAC获得Zunyimycins等系列具有很好抗MRSA活性新卤化抗生素,表明这些方法的组合不失为一种有效的抗生素开发手段[12-14]。我国科学家在Ⅱ型聚酮的发现、生物合成机制及调控机制解析等方面取得了巨大成就,特别是张立新、向文胜和王为善等科学家提出了“精准动态调控内源三酰甘油(TAGs)提高聚酮产量工程策略”[25]被认为“这是70年来首次在代谢水平上清晰阐明链霉菌初级代谢到次级代谢的代谢转换机制并进行工程应用”,利用该策略已成功实现了土霉素、杰多霉素等Ⅱ型聚酮的高产菌株的构建,给低产抗生素的开发提供一条新的策略。
3.2 基于基因操作的放线菌活性天然产物开发
延安市微生物药物创新及转化重点实验室利用次级代谢基因簇在线分析软件antiSMASH[26]对天蓝色链霉菌A3(2)基因组完成图序列 (GenBank No.NC_003888.3)进行了次级代谢产物基因簇预测。结果表明,该基因组中至少蕴藏着25个独立的次级代谢产物生物合成基因簇,这些基因簇在不同的条件下可能产生一系列的结构类似物,极大地丰富了天蓝色链霉菌的次级代谢产物产生潜力。截至2014年,已报道在天蓝色链霉菌A3(2)中发现了17种天然产物[27],分析统计结果表明,目前已有14类次级代谢合成途径编码基因簇在天蓝色链霉菌A3(2)中找到了与之相对应的次级代谢产物,也就是说,经过众多科学家多年的努力,在链霉菌的模式生物中尚有11种基因簇未能找到与之相对应的次级代谢产物。这更进一步说明,链霉菌的次级代谢潜力远远高于人们以前的预期 (即除了部分基因簇对应于已知的活性天然产物外,尚有部分代谢途径还没有相应的天然产物与之对应)。Hsiao等利用天蓝色链霉菌A3(2)的次级代谢相关的基因芯片分析了阿维链霉菌(Streptomycesavermitilis)、卡特兰链霉菌(Streptomycescattleya)、海洋链霉菌(Streptomycesmaritimus)和金黄色北孢菌(Kitasatosporaaureofaciens)等4株菌的基因组后,发现至少有23个次级代谢基因簇相关基因在这4株菌中均能找到[28],但从后3株菌中找到的相应基因簇编码合成的产物数量远少于天蓝色链霉菌A3(2)。科学家们开始着手对这些基因簇进行深入的开发,期待对这些基因簇的激活挖掘能发现新的次级代谢产物。
放线菌次级代谢产物生物合成基因簇在实验室培养环境的生理状态下,往往是以沉默状态存在,而且对于很多即使以活性状态存在的次级代谢基因簇而言,其表达水平往往很低,这就给其相应的产物纯化和鉴定带来很大困难。由于隐性基因簇可能编码合成具有成药潜力的新型化合物,因此激活或提高次级代谢隐性基因簇的表达水平对于发现新的次级代谢产物具有极大意义。人们将利用各种手段激活沉默基因簇的表达或者提高其表达水平的操作概称为基因组挖掘(genome mining)。目前,常见的微生物基因组挖掘手段主要有抗终止、途径特异性调控激活、基因簇倍增、组合生物合成、代谢工程等[29]。近年来发展的CRISPR-BEST[30]或aMSGE[31]等对相应的蛋白编码基因或调控序列进行遗传操作也极大地提高了发现放线菌来源新抗生素的机会。
基于多拷贝基因簇可以大幅度提高编码天然产物合成量,研究者可以利基因编辑技术,将完整的生物合成基因簇克隆且定向定点地整合到放线菌基因组特定位点,为该产物高效生物合成创造条件。Liao等[32]将完整的Nikkomycin生物合成基因簇整合到产生菌异色链霉菌(Streptomycesansochromogenes) 7100基因组上,成功地获得Nikkomycin高产菌株 (产量由340 mg/L提高到1 100 mg/L)。
组合生物合成 (Combinatorial biosynthesis)是在人们克隆得到大量的微生物次级代谢产物生物合成基因簇及对相关合成基因功能进行分析,以及了解其生物合成、调节等基因的基础上,通过基因编辑等DNA操作技术在体外对这些不同来源的基因进行删除、倍增、添加、取代以及重组,然后导入到一个适当的微生物宿主中并定向合成新抗生素或其他一些新的生理活性物质的一种复杂、综合性生物技术[33]。Basnet等[34]将clorobiocin的卤化酶基因与novobiocin生物合成基因簇在天蓝色链霉菌M152中共表达,并将甲基化酶NovO阻断,成功获得卤化取代甲基的新卤化物noclobiocin114。 Basnet等[34]将委内瑞拉链霉菌(Streptomycesvenezuelae)来源的P450编码基因pikC导入到erythromycin生物合成基因簇的eryBV编码基因删除的红糖多孢菌(Saccharopolysporaerythraea)菌株中,进行异源表达获得了与Erythromycin B具有相似的生物活性的新结构类似物5-O-desosaminyl erythronolide B 和5-O-desosaminyl erythronolide A。Kunakom等[35]也关注到了Alessandra等将clorobiocin的卤化酶基因与novobiocin生物合成基因簇的在天蓝色链霉菌M152中共表达,并将该基因簇的甲基化酶NovO阻断,成功获得了糖基C3氨基被吡咯取代的新型化合物noclobiocin102。Han等[36]分别将参与糖基合成desIII、desIV、desI、desII、desV、desVI等基因与desVII/desVIII进行不同基因间组合整合入ermE启动子调控下的pSET34质粒载体,构建了一系列的重组穿梭质粒载体,并分别转化敲除了糖基合成基因簇的委内瑞拉链霉菌YJ003菌株,成功得到了正常的天然产物narbomycin及系列的衍生物。Minami等[37]利用参与次级代谢糖基化酶基因不保守性和识别糖基配体的宽容性合成新型糖基化修饰原理, 将Vicenistatin糖基化酶异源表达纯化酶蛋白催化一系列的配醣体,得到了dTDP-vicenisamine修饰的系列衍生物。Myronovskyi等[38]将不同来源的糖基配体合成基因簇与糖基化酶基因的组合生物合成,通过异源组合表达成功实现angucyclines、aranciamycins等新化合物的系列突变衍生物合成。
4 展 望
尽管特殊生境来源放线菌资源收集结合生理水平和基因水平的天然产物挖掘已经极大地增加了研究者通过基因组挖掘的方法获得新次级代谢产物的机会,促进了放线菌活性天然产物开发,但是,由于开发抗耐药菌感染抗生素的过程异乎寻常地困难,很多研究者和制药公司放弃了这方面的研究,2011 年辉瑞制药关闭了其抗生素研发中心,也意味着抗生素开发泥土时代的落幕。目前,全球前18家最大的制药公司中,已有15家彻底退出了抗生素市场,抗生素的开发前景这些问题被提上日程。后新冠肺炎疫情时代,由于SARS-Cov2可能在很长的时间内将和人类社会共存,越来越多的证据表明,大量新冠肺炎患者在住院期间被诊断出患有继发感染[39]。目前尚不清楚这些感染的具体原因,但有迹象表明,患有病毒性呼吸道感染的患者更易出现细菌感染,引起这些继发感染的细菌是对多种药物具有耐药性的细菌[40]。后疫情新冠肺炎时代, 开发抗耐药菌感染的新抗生素更应该引起人们的重视。
抗耐药菌抗生素的开发面临诸多的技术瓶颈,针对耐药靶点的高通量药物筛选等技术的进步给包括放线菌来源的抗耐药菌抗生素的开发带来巨大的机遇[41-42],新抗生素的开发不再是对一个商业化的产物库的简单的筛选和抗菌效果验证问题,而是需要充分考虑多方面技术综合集成的多学科的充满挑战的工作[42-43]。将微生物表观遗传等技术引入放线菌天然产物开发,抗生素痕量分析技术引入活性天然产物生物合成的检测,天然产物结构解析平台及软件的改进、代谢中间产物示踪技术的发展、基因编辑及代谢工程的组合应用[44-49]等可能会给放线菌来源抗耐药菌感染抗生素带来新的明天[50-51]。
致谢感谢陕西省自然科学础研究计划面上项目(2020JM-545,2020JM-550,2021JM-416)、大学生创新创业训练计划项目(D2019224,D2020146,D2020167,D2020161,202110719023)对本工作的支持。尚坤、阎瑾逸与高君梅对本工作有同等贡献。

